Dr Mateusz Wilamowski z Zakładu Biochemii Ogólnej pierwszym autorem publikacji opisującej metylację czapeczki RNA w SARS-CoV-2, która ukazała się w renomowanym czasopiśmie „Proceedings of the National Academy of Sciences of the United States of America”.
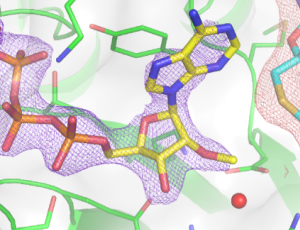
Jednym z kompleksów białkowych SARS-CoV-2 jest heterodimer Nsp10/Nsp16 o aktywności 2'-O-metylotransferazy. Dr Mateusz Wilamowski i inni naukowcy stosując technikę seryjnej krystalografii rozwiązali strukturę kryształu Nsp10/16 wraz z czapeczką RNA metylowaną w pozycji 2′-O rybozy (m7GpppAm2′-O). Aktywność Nsp10/16 z SARS-CoV-2 jest wymagana do metylacji czapeczki wirusowego RNA na końcu 5' transkryptu. Przeniesienie grupy metylowej jest zależne od S-adenozylo-L-metioniny (AdoMet), służącej jako donor grupy metylowej. Grupa metylowa z AdoMet zostaje przesunięta do pozycji 2′-O rybozy adenozyny w cząsteczce Cap-0 w celu utworzenia produktów reakcji: Cap-1 i AdoHcys. Metylacja 2′-O czapeczki RNA pozwala SARS-CoV-2 naśladować transkrypty gospodarza, a tym samym oszukać mechanizmy odporności wrodzonej człowieka, które rozpoznają wirusowe/egzogenne RNA następnie aktywując odpowiedź immunologiczną zależną od interferonów.
Wyniki opisane w publikacji pt. „2′-O methylation of RNA cap in SARS-CoV-2 captured by serial crystallography”, są rezultatem pracy naukowców z Laboratorium Argonne, Uniwersytetu w Chicago, oraz Uniwersytetu Northwestern. Doktor Wilamowski w trakcie realizacji eksperymentów uczestniczył w stażu podoktorskim na Uniwersytecie Chicagowskim w grupie prof. Andrzeja Joachimiaka.
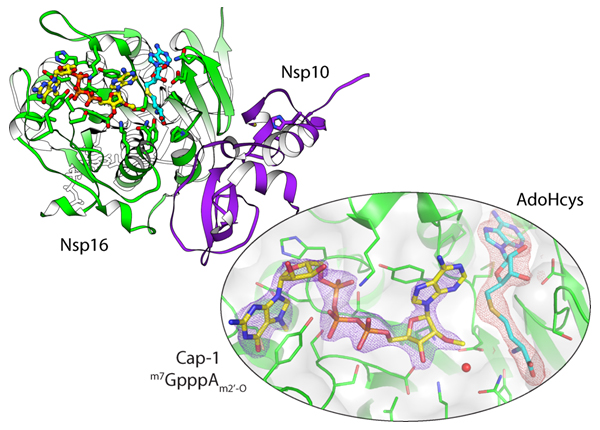
Analizy krystalograficzne wykonano z zastosowaniem unikalnych urządzeń badawczych synchrotronu Advanced Photon Source (APS) oraz superkomputera Theta w Laboratorium Argonne w Stanach Zjednoczonych. Wyniki opisane w pracy pokazują pierwsze struktury kryształów białek SARS-CoV-2 rozwiązane techniką seryjnej krystalografii. Publikacja w PNAS opisuje 4 struktury kompleksu Nsp10/16 z substratami i produktami katalizowanej reakcji – transferu grupy metylowej. Dane krystalograficzne zostały zgromadzone w APS z dziesiątek tysięcy mikrokryształów Nsp10/16 w temperaturze pokojowej. Metodyka seryjnej krystalografii pozwala uniknąć efektu uszkodzeń radiacyjnych czyniąc uzyskane wyniki bliższymi stanowi fizjologicznemu.
Pomimo rozpowszechnienia programu szczepień efektywne zwalczanie skutków SARS-CoV-2 wymaga leku, który może zostać podany pacjentom chorym na COVID-19. Cząsteczki, które wpływają na aktywność Nsp10/16, a tym samym hamują dojrzewanie wirusowego RNA, są potencjalnymi lekami przeciwko COVID-19, czyniąc SARS-CoV-2 podatnym na wczesną aktywację wrodzonej odpowiedzi immunologicznej. Pokazane w pracy w PNAS struktury kryształów Nsp10/16 oprócz wyjaśnienia podstawowych mechanizmów aktywności kompleksu 2'-O-metylotransferazy mogą zostać wykorzystane w celu inteligentnego projektowania inhibitorów z zastosowaniem metod modelowania molekularnego.
Finansowanie: National Institute of Allergy and Infectious Diseases, National Institutes of Health, Department of Health and Human Services, Contract HHSN272201700060C. DOE Office of Science through the National Virtual Biotechnology Laboratory, a consortium of DOE national laboratories focused on response to COVID-19, with funding provided by the CARES Act. The use of SBC beamlines at the Advanced Photon Source and Argonne Leadership Computing Facility is supported by the U.S. Department of Energy (DOE) Office of Science and operated for the DOE Office of Science by Argonne National Laboratory under Contract No. DE-AC02-06CH11357.

